一步步教你利用Blast分析驗(yàn)證引物序列
引物設(shè)計(jì)完成之后,需要用軟件 進(jìn)行分析,找到最佳的引物對(duì),采用Blast分析驗(yàn)證引物,可以知道序列的正確性,也能知道引物的特異性狀況、引物的具體位置以及PCR產(chǎn)物的大小。
先找到需要設(shè)計(jì)引物的目標(biāo)基因的在有引物序列的mRNA序列,可以通過(guò)全文(如最先發(fā)現(xiàn)該基因的文章),全文中有時(shí)可以找到大鼠c-jun mRNA序列的ACCESSION NUMBER,在PubMed中的“Nucleotide”中用此ACCESSION NUMBER就能找到序列。
可以利用這個(gè)ACCESSION NUMBER在PubMed中的“Nucleotide”中搜索到序列后,點(diǎn)擊右邊的“Links”,再點(diǎn)擊里面的“Related Sequences”就能找到所有的大鼠c-jun mRNA序列及其他物種 的一些c-jun mRNA序列。
文獻(xiàn)中的引物最好先驗(yàn)證其序列正確,并用軟件 分析引物比較好后再采用。通過(guò)BLAST驗(yàn)證,既可知道序列是否正確,也可同時(shí)了解引物的特異性、引物的位置及PCR產(chǎn)物的大小等。
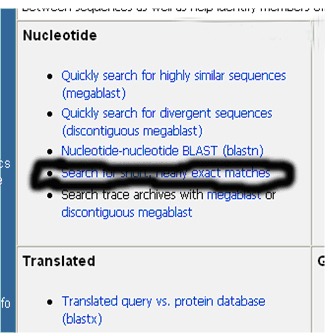
如果僅僅是為了驗(yàn)證引物序列是否正確(僅適合于基于完全匹配原則設(shè)計(jì)的引物),也可以用Blast的“Nucleotide-nucleotide BLAST (blastn )”或“Search for short, nearly exact matches”搜索,具體方法為:
1. 打開Blast,點(diǎn)擊“Nucleotide-nucleotide BLAST (blastn)”或“Search for short, nearly exact matches”
2. 等新頁(yè)面完全顯示后,將引物序列直接copy到“Search”框(沒(méi)有必要將下游引物序列轉(zhuǎn)換成其互補(bǔ)鏈),下面3種方法都可以(我覺(jué)得3種方法的搜索結(jié)果沒(méi)什么區(qū)別,沒(méi)仔細(xì)比較過(guò)哦!)
(1)直接拷貝上游引物序列,然后直接將下游引物序列拷貝在上游引物后面
(2)直接拷貝上游引物序列,在上游引物序列后加一空格,然后直接將下游引物序列拷貝在空格后面
(3)直接拷貝上游引物序列,換行,然后直接拷貝下游引物序列
注意:記住上、下游引物的長(zhǎng)度,如上游引物為19bp、下游引物為18bp,這在查看Blast結(jié)果時(shí)有用。
3. 點(diǎn)擊“BLAST!”,新頁(yè)面完全出來(lái)以后,點(diǎn)擊“Format!”。
4. 結(jié)果完全出來(lái)后,查找有沒(méi)有和1-19、20-37同時(shí)完全匹配的基因,其他的就不用考慮了。當(dāng)然,如果下游引物序列所對(duì)應(yīng)的基因片段的
3'端正好和上游引物序列的3'端的1或2個(gè)堿基互補(bǔ),那就可能是19-37或18-37。
如果有,那你的引物序列就是正確的。從文獻(xiàn)上查的引物,除了要用Blast驗(yàn)證其序列是否正確外,還是應(yīng)該用軟件 分析分析到底引物好不好。
實(shí)例
1、進(jìn)入網(wǎng)頁(yè):
http://www.NCBI .nlm.nih.gov/BLAST/
2、點(diǎn)擊 Search for short, nearly exact matches
3、 在search欄中輸入引物系列:
注:文獻(xiàn)報(bào)道ABCG2的引物為
5’-CTGAGATCCTGAGCCTTTGG-3’;
5’-TGCCCATCACAACATCATCT-3’
(1) 輸入方法可先輸入上游引物,進(jìn)行blast程序,同樣方法在進(jìn)行下游引物的blast程序。
這種方法較繁瑣,而且在結(jié)果分析特異性時(shí)要看能與上游引物的匹配的系列,還要看與下游引物匹配的系列——之后看兩者的交叉。
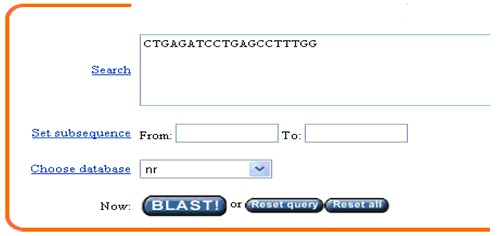
(2) 簡(jiǎn)便的做法是同時(shí)輸入上下游引物:有以下兩種方法。輸入上下游引物系列都從5’——3’。
A、輸入上游引物 空格 輸入下游引物

B、輸入上游引物 回車 輸入下游引物

4、 在options for advanced blasting中:select from 欄選擇 Homo sapiens【ORGN】,Expect后面的數(shù)字改為10
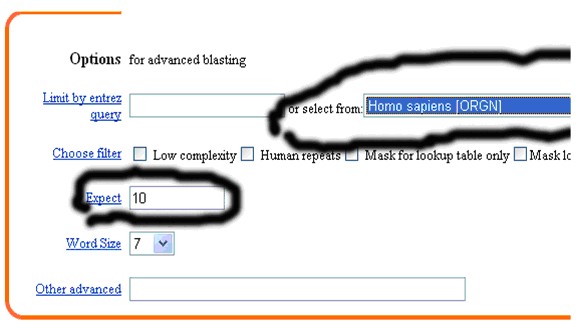
5、在format中:select from 欄選擇Homo sapiens【ORGN】,Expect后面的數(shù)字填上0 10。

6、 點(diǎn)擊網(wǎng)頁(yè)中最下面的“BLAST!”

7、 出現(xiàn)新的網(wǎng)頁(yè),點(diǎn)擊Format!
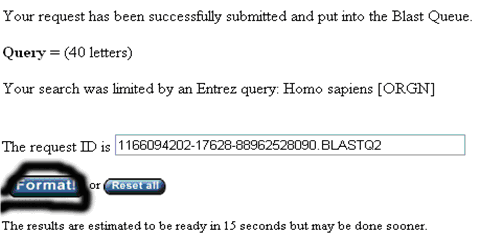
8、 等待若干秒之后,出現(xiàn)results of BLAST的網(wǎng)頁(yè)。該網(wǎng)頁(yè)用三種形式來(lái)顯示blast的結(jié)果。
(1)圖形格式:

圖中①代表這些序列與上游引物匹配、并與下游引物互補(bǔ)的得分值都位于40~50分
圖中②代表這些序列與上游引物匹配的得分值位于40~50分,而與下游引物不互補(bǔ)
圖中③代表這些序列與下游引物互補(bǔ)的得分值小于40分,而與上游引物不匹配
通過(guò)點(diǎn)擊相應(yīng)的bar可以得到匹配情況的詳細(xì)信息。
(2)結(jié)果信息概要:

從左到右分別為:
A、數(shù)據(jù)庫(kù)系列的身份證:點(diǎn)擊之后可以獲得該序列的信息
B、系列的簡(jiǎn)單描述
C、高比值片段對(duì)(high-scoring segment pairs, HSP)的字符得分。按照得分的高低由大到小排列。得分的計(jì)算公式=匹配的堿基×2+0.1。舉例:如果有20個(gè)堿基匹配,則其得分為40.1。
D、E值:代表被比對(duì)的兩個(gè)序列不相關(guān)的可能性。【The E value decreases exponentially as the Score (S) that is assigned to a match between two sequences increases】。E值最低的最有意義,也就是說(shuō)序列的相似性最大。設(shè)定的E值是我們限定的上限,E值太高的就不顯示了
E、最后一欄有的有UEG的字樣,其中:
U代表:Unigene數(shù)據(jù)庫(kù)
E代表:GEO profiles數(shù)據(jù)庫(kù)
G代表:Gene數(shù)據(jù)庫(kù)
(3)結(jié)果詳細(xì)信息:

① 圈出來(lái)的部分代表序列的信息
② 第一個(gè)大括號(hào)代表上游引物與該序列的正鏈【Plus/Plus】的匹配情況:
共有21個(gè)堿基匹配,得分42.1分【21×2+0.1=42.1】,E值為0.020
上游引物與序列的2143~2163位點(diǎn)匹配
③ 第二個(gè)大括號(hào)代表下游引物與該序列的負(fù)鏈【Plus/Minus】的匹配情況:
共有20個(gè)堿基匹配,得分40.1分【20×2+0.1=40.1】,E值為0.077。
下游引物與該序列的29360~29379位點(diǎn)互補(bǔ)
注意點(diǎn):
①上游引物為20個(gè)堿基,為什么會(huì)變成21個(gè)堿基呢?這是因?yàn)橄掠我锏牡谝粋€(gè)堿基為T,剛好與系列的2163位點(diǎn)的T匹配,因此下游引物的開頭的第一個(gè)堿基被當(dāng)成了上游引物了。同理,上游引物的最后一個(gè)堿基為G,被當(dāng)成了下游引物了。通過(guò)尋找有沒(méi)有與1~20位點(diǎn)、20~40位點(diǎn)完全匹配的序列,就可以避免這個(gè)因素的干擾了。
②為什么與上下游引物匹配的ABCG2序列有多種
A、 為同一個(gè)基因來(lái)源的不同的mRNA片段
B、 為該基因的DNA系列
C、 為同一個(gè)基因來(lái)源的不同的cDNA克隆片段
結(jié)果判斷:
①驗(yàn)證文獻(xiàn)報(bào)道的引物是否正確:如果你可以在所顯示的結(jié)果中找出你的目的基因,一般說(shuō)明你的引物正確性沒(méi)問(wèn)題。如果你blast后沒(méi)有發(fā)現(xiàn)你的目的基因,或者分值很低,該引物就可能不適合用。
②檢測(cè)該引物是否存有同源序列相匹配,造成PCR反應(yīng)的非特異擴(kuò)增。如果上游引物和下游引物都找到的相同的基因,并且分?jǐn)?shù)非常高。這種結(jié)果表明所設(shè)計(jì)的引物序列特異性不高,最好重新設(shè)計(jì)引物,否則會(huì)得到非特異擴(kuò)增的PCR產(chǎn)物。
北京天優(yōu)福康生物科技有限公司
官網(wǎng):http://www.njjinzhong.com/
服務(wù)熱線:400-860-6160
聯(lián)系電話/微信:13718308763
QQ:2136615612 3317607072
E-mail:Tianyoubzwz@163.com


